Los aminoácidos
Los aminoácidos se llaman así porque son derivados aminados de ácidos carboxílicos. En los 20 aminoácidos comunes, los grupos amino y carboxilo están unidos al mismo átomo de carbono: (carbono alfa) . Así, todos los aminoácidos estándar que contienen las proteínas son a-aminoácidos. Al carbono alfa se unen otros dos sustituyentes: un átomo de hidrógeno y una cadena lateral (R) que es única para cada aminoácido. En los nombres químicos de los aminoácidos, los átomos de carbono se identifican con números que comienzan en el átomo de carbono del grupo carboxilo.
Estructura
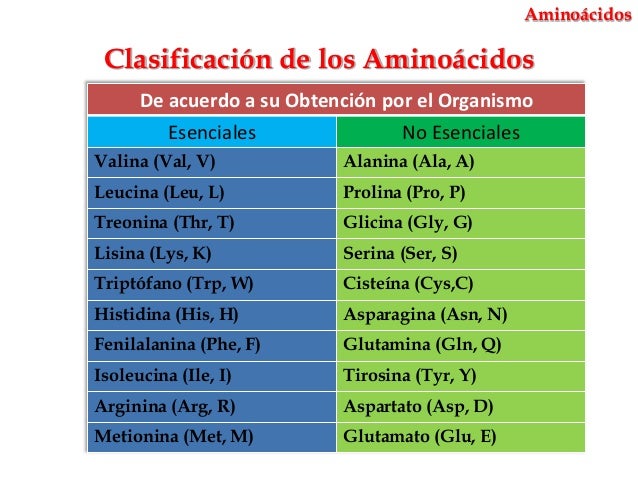
Clasificación 1: de acuerdo a su obtencion por el organismo
Clasificación 2: de acuerdo a sus cadenas laterales

Clasificación 3: de acuerdo a su hidropatía
- Muy hidrofóbico: Isoleucina, Fenilalanina, Valina, Leucina, Metionina,
- Menos hidrofóbicos: Triptófano, Alanina, Glicina, Cisteína, Tirosina, Prolina, Treonina, Serina.
- hidrofílico: Histidina, Glutamato, Asparagina, Glutamina, Aspartato, Lisina, Arginina.
Estereoquímica
En 19 de los 20 aminoácidos que se usan en la biosíntesis de proteínas, el átomo de carbono alfa es quiral, o asimétrico, porque tiene cuatro grupos diferentes unidos a él.
La excepción es la glicina, cuyo grupo R sólo es un átomo de hidrógeno (la molécula no es quiral, porque el átomo de carbono a está unida a dos átomos idénticos de hidrógeno).
Los 19 aminoácidos quirales pueden, en consecuencia, existir como estereoisómeros. Los estereoisómeros son compuestos que tienen la misma fórmula molecular pero difieren en el orden o configuración de sus átomos en el espacio.
Los dos estereoisómeros son moléculas distintas que no se pueden interconvertir con facilidad entre sí ya que un cambio de configuración requiere romper uno o más enlaces. Los estereoisómeros de aminoácidos no son imágenes especulares (es decir, imágenes en espejo) superponibles; a dichos estereoisómeros se les llama enantiómeros.
ejemplo:
Dos de los 19 aminoácidos quirales, la isoleucina y la treonina, presentan dos átomos quirales de carbono cada uno. La isoleucina y la treonina pueden formar, cada una, cuatro estereoisómeros diferentes.

Propiedades
ionizacion de los aminoácidos
Las propiedades físicas de los aminoácidos reciben influencias de los estados iónicos de los grupos a-carboxilo y a-amino y de todos los grupos ionizables que haya en las cadenas laterales. Cada grupo ionizable guarda relación con un valor específico de pKa, que corresponde al pH al que son iguales las concentraciones de las formas protonada y no protonada.
Cuando el pH de la solución es menor que el pKa, predomina la forma protonada y el aminoácido es entonces un ácido real, capaz de donar un protón. Cuando el pH de la solución es mayor que el pKa del grupo ionizable, la forma no protonada de ese grupo predomina, y el aminoácido existe en forma de base conjugada, que es aceptora de protones.
Cada aminoácido tiene al menos dos valores de pKa que corresponden a la ionización de los grupos a-carboxilo y a-amino. Además, siete de los aminoácidos comunes tienen cadenas laterales ionizables con valores adicionales y medibles de pKa. Esos valores difieren entre los aminoácidos. Así, a determinado pH, con frecuencia los aminoácidos tienen cargas netas diferentes.
Los estados iónicos de las cadenas laterales de los aminoácidos influyen sobre las estructuras tridimensionales de las proteínas. Además, ya que varios residuos ionizables de aminoácido intervienen en catálisis por enzimas, la comprensión de las propiedades iónicas de los aminoácidos ayuda a comprender los mecanismos enzimáticos.
Método de análisis de mezclas de aminoácidos
- Cromatografía en columna
 funciona para fraccionar la mezcla de proteínas que resta después de la precipitación con sulfato de amonio y la diálisis. Una columna cilíndrica se llena con un material insoluble, como fibras de celulosa sustituida o esferillas de material sintético. La mezcla de proteínas se agrega a la columna y se lava haciendo pasar por la matriz de material insoluble un solvente. A medida que el solvente fluye por la columna, el eluido (que es el líquido que sale por el fondo de la columna) se recolecta en muchas fracciones. La velocidad con la que las proteínas atraviesan la matriz depende de las interacciones entre matriz y proteína.
funciona para fraccionar la mezcla de proteínas que resta después de la precipitación con sulfato de amonio y la diálisis. Una columna cilíndrica se llena con un material insoluble, como fibras de celulosa sustituida o esferillas de material sintético. La mezcla de proteínas se agrega a la columna y se lava haciendo pasar por la matriz de material insoluble un solvente. A medida que el solvente fluye por la columna, el eluido (que es el líquido que sale por el fondo de la columna) se recolecta en muchas fracciones. La velocidad con la que las proteínas atraviesan la matriz depende de las interacciones entre matriz y proteína.- Cromatografía de intercambio iónico
la matriz tiene cargas positivas (resinas de intercambio de aniones) o cargas negativas (resinas de intercambio de cationes). Las matrices de intercambio aniónico se unen con proteínas con carga negativa y las retienen para su posterior elución. Por el contrario, los materiales de intercambio catiónico se enlazan con proteínas con carga positiva. Las proteínas enlazadas se pueden eluir en serie si se aumenta la concentración salina del solvente en forma gradual. A medida que aumenta la concentración de la sal, llega a una concentración en la que sus iones superan a las proteínas en la unión a la matriz. A esa concentración la proteína se libera y es recolectada en el eluido. Las proteínas unidas individualmente se eluyen a distintas concentraciones de sal, y este fraccionamiento determina que la cromatografía de intercambio iónico constituya un método poderoso para purificar proteínas.
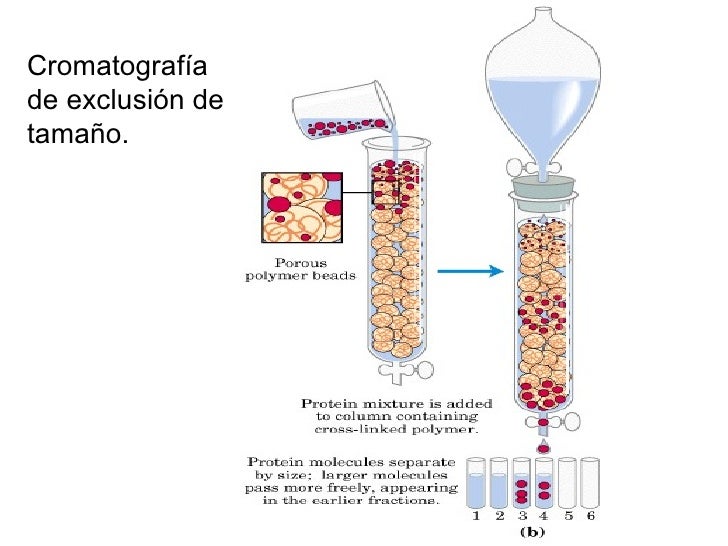
- cromatografía por filtración en gel
separa a las proteínas con base en su tamaño molecular. El gel es una matriz de esferillas porosas. Las proteínas que son menores que el tamaño promedio del poro penetran en gran parte del volumen interno de las perlas, y de ese modo se retardan por la matriz cuando la solución amortiguadora fluye por la columna. Mientras menor sea la proteína, se eluye con más retraso de la columna. Hay menos poros accesibles a las moléculas mayores de proteína. En consecuencia, las proteínas más grandes rodean a las perlas y se eluyen primero.
- cromatografía de afinidad
es el tipo de cromatografía en columna más selectivo. Se basa en interacciones específicas de unión entre la proteína deseada y alguna otra molécula enlazada en forma covalente a la matriz de la columna. La molécula unida a la matriz puede ser una sustancia o ligando que se une a una proteína in vivo; un anticuerpo que reconozca a la proteína deseada u otra proteína de la cual se conozca el modo de interacción con la proteína deseada dentro de la célula. A medida que la mezcla de proteínas pasa por la columna, sólo la proteína deseada se une en forma específica a la matriz. A continuación la columna se lava varias veces con solución amortiguadora para deshacerse de las proteínas adheridas en forma inespecífica. Por último, la proteína deseada puede eluirse lavando la columna con un solvente con alta concentración de sal, que rompa la interacción entre la proteína y la matriz de la columna. En algunos casos se puede extraer la proteína ligada en forma selectiva de la columna de afinidad agregando un exceso de ligando al amortiguador de elución. La proteína deseada se une
preferencialmente al ligando en la solución y no a la menor concentración de ligando que está unido a la matriz insoluble en la columna. Este método es más efectivo cuando el ligando es una molécula pequeña. La cromatografía de afinidad puede, por sí misma, purificar en ocasiones una proteína de 1000 a 10,000 veces.